Dplyr es quizás uno de los paquetes más importantes del Tidyverse pues permite manipular dataframes fácilmente en R, a través de funciones como mutate(), select(), filter(), summarise(), arrange() y group_by().
Cuando se usa con el operador pipe %>%, el código hecho con este paquete es muy legible, por lo que hay menos riesgos de olvidar cuál fue el procedimiento que aplicamos en nuestra base de datos.
Más allá de las funciones más comunes, hoy te quiero hablar de 3 funciones de dplyr que probablemente no conocías.
1. Transmute#
En el trabajo del día a día, muchas veces me encuentro con la necesidad de sacar estadísticas de resumen de un dataframe dado, y lo que suele pasar es que llamo la función mutate() para computar alguna cifra en específico (por ejemplo, la tasa de homicidios por cien mil habitantes usando los registros de homicidios y la población) y luego tengo que usar select() para dejar únicamente las columnas que me interesan en una tabla final.
Sin embargo, la función trasmute() puede ahorrarte estos pasos, pues permite crear nuevas variables en el dataframe y al tiempo borra las que no usaste.
Veamos un ejemplo. Al instalar dplyr también descargas un dataframe llamado starwars que incluye la información de 87 personajes que aparecen entre en Episodio I y el Episodio VII. Entre las características de los personajes se incluyen la altura, el peso, el sexo, el planeta en que nacieron, etc.
Supongamos que -por alguna razón- queremos calcular el Body Mass Index de todos los personajes. Si lo hicieramos usando mutate() y select() tendríamos que hacer algo de este estilo:
starwars %>% mutate(BMI = mass/(height/100)^2) %>%
select(name, height, mass, BMI)
Ahora bien, usando transmute(), podemos abreviar esta operación de la siguiente manera:
starwars %>% transmute(name, height, mass, BMI = mass/(height/100)^2)
Como puedes ver, sólo basta con llamar el nombre de una columna dentro de transmute() para que salga en la tabla final, y en la misma línea puedes crear nuevas variables, como si lo hicieras con la función mutate().
Y si, el personaje con mayor BMI es Jabba, con un valor de 443.
2. Coalesce#
¿Alguna vez te has encontrado con una tabla que quieras importar, en la que una columna esté separada en varias? Supón que tenemos una tabla con el resultado final de 5 estudiantes en una materia cualquiera. Sin embargo, en vez de anotar este resultado en una columna, la persona que hizo los registros los separó de la siguiente manera.
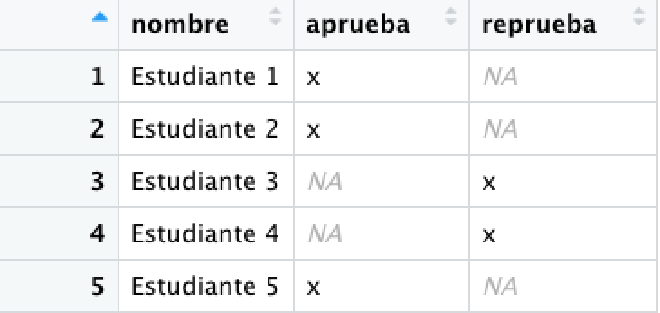
Para este tipo de casos, la función coalesce() funciona perfectamente. Esta función encuentra el primer valor no vacío para cada posición y también permite juntar en una sola columna los valores separados como en el ejemplo anterior. Para este caso, primero vamos a cambiar un poco el contenido de las columnas, de tal manera que no nos quede sólo una columna de x, y luego crearemos la nueva columna usando esta función.
resultados %>% mutate(aprueba = str_replace(aprueba, "x", "aprueba")) %>%
mutate(reprueba = str_replace(reprueba, "x", "reprueba")) %>%
mutate(resultado = coalesce(aprueba, reprueba))
Del código anterior nos quedará la siguiente tabla:

Aquí pudimos haber usado la función transmute() para que no quedaran las columnas aprueba y reprueba.
3. Slice#
Usualmente cuando tenemos que seleccionar algunas filas de un dataframe con dplyr, lo hacemos con la función filter(), que recibe como argumento un condicional. A modo de ejemplo, si queremos obtener los paises con un PIB per cápita superior a 3.000 USD en 2007 de la base de datos de gapminder, lo que haríamos es lo siguiente:
library(gapminder)
gapminder %>% filter(year == 2007, gdpPercap > 3000)
Sin embargo, no en todos los casos queremos sacar parte de las filas de esta manera. Así como en R base podemos obtener un subconjunto de filas usando el operador de extracción [], dplyr contiene la función slice(). A modo de ejemplo, supongamos que queremos obtener el país con mayor población para cada continente en 1972. Esto lo podemos hacer de la siguiente manera:
library(gapminder)
gapminder %>% filter(year == 1972) %>%
group_by(continent) %>%
arrange(continent, desc(pop)) %>%
slice(1)
Dado que había ordenado la tabla en función al continente y a la población, aplicar la función slice() devuelve directamente el país con mayor población por cada continente:
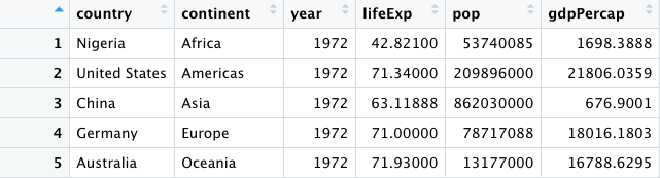
Como puedes ver, que además de permitir extraer filas con el número asociado a su posición, la función slice()se puede encadenar con el pipe %>% y funciona muy bien con funciones como group_by(). En este caso, en vez de devolver sólo una fila, devolvió una fila por cada uno de los grupos que definimos. En este caso, los continentes.
Bonus: Crucigrama de dplyr#
La inspiración de este post me llegó por un crucigrama que descrubrí hace algunas semanas en el blog de Georgios Karamanis sobre funciones de dplyr. En mi primer intento logré llenar sólo 15 de las 34 funciones sin mirar en la documentación. ¡Te invito a que resuelvas este crucigrama e intentes superar mi resultado!